[혼공머신러닝] 2 - 2 데이터 전처리
데이터 전처리
1. 용어 정리
스케일 : 두 특성의 값이 놓인 범위가 다를 때
표준점수 : 각 특성값이 평균에서 표준편차의 몇 배만큼 떨어져 있는지를 나타내는 것
브로드캐스팅 : 일정 조건을 부합하는 다른 형태의 배열끼리 연산을 수행하는 것을 의미합니다.
2. 문제 풀이
2 - 1. numpy로 데이터 준비하기
먼저 생선 데이터를 준비합니다.
fish_length = [25.4, 26.3, 26.5, 29.0, 29.0, 29.7, 29.7, 30.0, 30.0, 30.7, 31.0, 31.0,
31.5, 32.0, 32.0, 32.0, 33.0, 33.0, 33.5, 33.5, 34.0, 34.0, 34.5, 35.0,
35.0, 35.0, 35.0, 36.0, 36.0, 37.0, 38.5, 38.5, 39.5, 41.0, 41.0, 9.8,
10.5, 10.6, 11.0, 11.2, 11.3, 11.8, 11.8, 12.0, 12.2, 12.4, 13.0, 14.3, 15.0]
fish_weight = [242.0, 290.0, 340.0, 363.0, 430.0, 450.0, 500.0, 390.0, 450.0, 500.0, 475.0, 500.0,
500.0, 340.0, 600.0, 600.0, 700.0, 700.0, 610.0, 650.0, 575.0, 685.0, 620.0, 680.0,
700.0, 725.0, 720.0, 714.0, 850.0, 1000.0, 920.0, 955.0, 925.0, 975.0, 950.0, 6.7,
7.5, 7.0, 9.7, 9.8, 8.7, 10.0, 9.9, 9.8, 12.2, 13.4, 12.2, 19.7, 19.9]
넘파이로 임포트 한 후에 간단하게 2개의 리스트를 튜플로 전달을 해봅니다.
import numpy as np
np.column_stack(([1,2,3], [4,5,6]))
array([[1, 4],
[2, 5],
[3, 6]])
fish_length와 fish_weight를 합친 후에 5개의 데이터를 확인해 봅니다.
fish_data = np.column_stack((fish_length, fish_weight))
print(fish_data[:5])
[[ 25.4 242. ]
[ 26.3 290. ]
[ 26.5 340. ]
[ 29. 363. ]
[ 29. 430. ]]
np.concatenate()함수를 사용하여 타깃 데이터를 만들어 보겠습니다.
fish_target = np.concatenate((np.ones(35), np.zeros(14)))
print(fish_target)
[1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1.
1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0.
0.]
2 - 2. 사이킷 런으로 훈련세트 테스트세트 나누기
넘파이를 대신하여 사이킷 런으로 훈련세트와 테스트 세트를 더 간단하게 할 수 있다는걸 배웠습니다. 이번에는 사이킷 런으로 분리를 해보겠습니다.
from sklearn.model_selection import train_test_split
train_input, test_input, train_target, test_target = train_test_split(
fish_data, fish_target, random_state=42)
train_test_split()함수는 자체적으로 시드를 지정할 수 있습니다. 이 함수는 기본적으로 25%를 테스트 세트로 떼어 냅니다. 아직은 비율이 정확하지 않아 stratify를 사용하여 클래스 비율에 맞게 데이터를 나눕니다.
train_input, test_input, train_target, test_target = train_test_split(
fish_data, fish_target, stratify=fish_target, random_state=42)
아직은 도미와 빙어를 완벽하게 구분 짓지는 못합니다. 이 문제를 해결하기 위해 kneighbors() 메서드에서 반환한 distances를 사용하여 이웃 샘플까지의 거리를 구해보겠습니다.
print(distances)
[[ 92.00086956 130.48375378 130.73859415 138.32150953 138.39320793]]
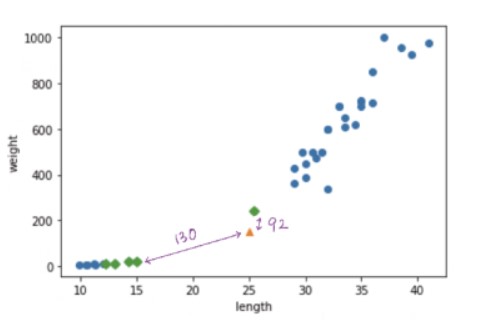
그림으로 확인을 해보면 거리의 비율이 이상하다는걸 알 수가 있습니다. x축의 범위가 좁고 y축의 범위가 넓기 때문에 이러한 오류가 생기는 겁니다.
2 - 3. 데이터 전처리
가장 널리 사용하는 전처리 방법 중 하나는 표준점수(z 점수라고도 불립니다.)입니다. 이를 통해 실제 특성값의 크기와 상관없이 동일한 조건으로 비교할 수 있습니다.
mean = np.mean(train_input, axis=0) #평균을 계산
std = np.std(train_input, axis=0) #표준편차를 계산
print(mean, std)
[ 27.29722222 454.09722222] [ 9.98244253 323.29893931]
평균과 표준편차를 표준점수로 반환하기
train_scaled = (train_input - mean) / std
2 - 4. 전처리 데이터로 모델 훈련하기
new = ([25, 150] - mean) / std
plt.scatter(train_scaled[:,0], train_scaled[:,1])
plt.scatter(new[0], new[1], marker='^')
plt.xlabel('length')
plt.ylabel('weight')
plt.show()
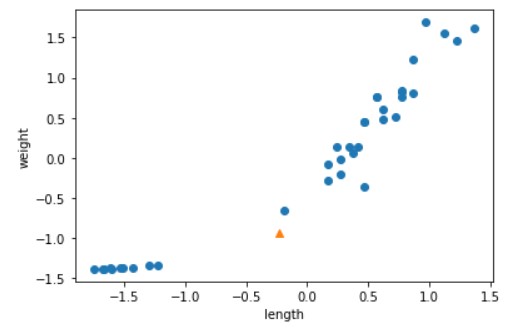
위와 같이 크게 달라진 점은 x축과 y축의 범위가 -1.5 ~ 1.5 사이로 바뀐것을 확인해 볼 수 있습니다. 다시한번 모델을 평가하면 완벽하게 분류가 된 것을 확인해 볼 수 있습니다.
test_scaled = (test_input - mean) / std
kn.score(test_scaled, test_target)
1.0
공부한 전체 코드는 깃허브에 올렸습니다. https://github.com/mgskko/Data_science_Study-hongongmachine/blob/main/%ED%98%BC%EA%B3%B5%EB%A8%B8%EC%8B%A0_2%EA%B0%95_2.ipynb

댓글남기기